La entrada anterior de la serie describió una de las técnicas maestras en los laboratorios de biología molecular (y otros) de todo el mundo: la PCR. Conviene tener bien claro el concepto porque vamos a comentar un par de cosas más al respecto.
Por ejemplo, nos centramos sólo en explicar cómo amplificar DNA. ¿Qué ocurre cuando partimos de moléculas de RNA purificado, que son de cadena sencilla? El proceso en cadena descrito anteriormente era válido para las moléculas de doble cadenaNo hay problema: volvemos a aplicar enzimas disponibles en la naturaleza para solucionarlo, en este caso la transcriptasa inversa (o retrotranscriptasa) víricas. No es más que una polimerasa particular que utiliza RNA como molde. El procedimiento para amplificar RNA más común es el siguiente: ponemos en el mismo tubo cierta cantidad de RNA purificado junto con secuencias cortas de muchas timinas (poliT); estos oligonucleótidos (...TTTTT...) se hibridan a alta temperatura (para lo cual utilizamos también un termociclador) con la colita de adeninas (---AAAA---) que tienen las moléculas de RNA sintetizadas en el núcleo (colita que las permite salir al citoplasma para la transcripción sin que las degraden). El papel de la poliT es análogo al de los cebadores para amplificar ambas cadenas del DNA. Tras la hibridación con las poliT, basta añadir a la reacción la trancriptasa inversa (más cara que la convencional) con su respectivo buffer o tampón, nucleótidos y completar el volumen con agua. Se deja actuar la transcriptasa inversa (o reversa) a durante una hora a la temperatura adecuada y se obtienen como resultado grandes cantidades de moléculas de cadena sencilla, equivalentes al RNA inicial, pero con timina en lugar de uracil. Es decir, similar al DNA de toda la vida pero sin intrones (secuencias no codificantes) y de cadena sencilla.
Esta molécula creada ex profeso en laboratorio se conoce como cDNA (DNA copia), y es una manera inmejorable de obtener una secuencia codificante de DNA (gen) sin presencia de intrones, que se puede purificar y usar para mil técnicas posteriores más (algunas de las cuales explicaremos dentro de poco).
El cDNA obtenido puede servir a su vez de base para una amplificación concencional de DNA: en la primera reacción como es obvio sólo se puede amplificar la cadena que existe, pero la cadena sencilla vieja formará una doble cadena con la nueva y en las reacciones siguientes partimos de la situación normal: moléculas de doble cadena listas para amplificar.
Esta reacción descrita se llama RT-PCR (PCR de transcriptasa reversa), no confundir con la PCR a tiempo real (Real Time PCR), sistema que sirve para cuantificar ácidos nucleicos (DNA y RNA).
Es una técnica más compleja que requiere un termociclador especial, capaz de medir fluorescencia. La idea es intercalar en la cadena del ácido nucleico una molécula que de señal fluorescente (fluoróforo), que se pueda detectar y cuantificar y cuya señal sea proporcional a la cantidad de producto de la PCR.
Existen variaciones y complicaciones en la técnica pero lo más habitual es detectar específicamente una secuencia utilizando oligonucleótidos marcados con fluoróforos (sonda), que contienen el fluoróforo en un extremo y una molécula secuestradora de la florescencia del fluoróforo en el otro (quencher). El contenido de la reacción de PCR es similar al habitual, también existen cebadores de inicio de la amplificación. La sonda es complementaria a un fragmento de la secuencia diana, pero la polimerasa amplifica a partir del cebador.
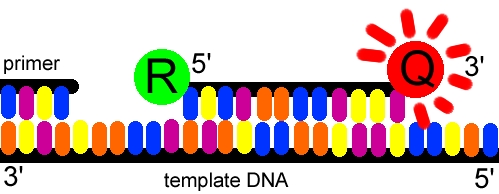 Cuando la polimerasa se tope con la sonda, separará el floróforo del resto de la sonda y se podrá detectar su fluorescencia (libre del quencher secuestrador), que aumentará exponencialmente según el número de copias del DNA molde. Se pueden usar varias sondas con fluoróforos distintos a la vez.
Cuando la polimerasa se tope con la sonda, separará el floróforo del resto de la sonda y se podrá detectar su fluorescencia (libre del quencher secuestrador), que aumentará exponencialmente según el número de copias del DNA molde. Se pueden usar varias sondas con fluoróforos distintos a la vez.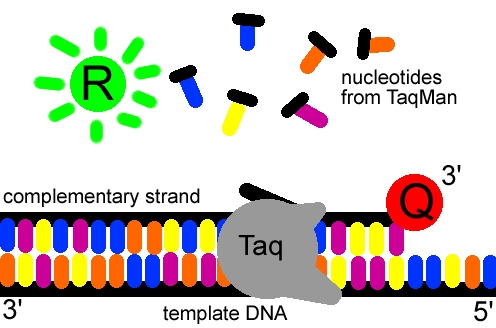
Para la cuantificación se analiza la curva de amplificación, que consta de 3 fases: una en la que el producto aún no se puede detectar, la fase de acumulación exponencial del producto y la fase de saturación de la reacción. El número de ciclos necesarios para que se produzca un aumento de fluorescencia significativo con respecto a la señal basal (ciclo umbral) es inversamente proporcional a la cantidad inicial de moléculas molde. Mediante los cálculos pertinentes (ya sería muy prolijo explicarlo) se puede obtener una cuantificación precisa del número de moléculas iniciales en la reacción.
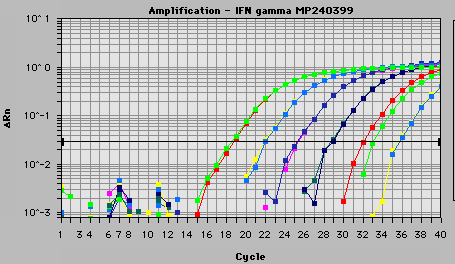
Os podeis hacer idea de la sofisticación creciente de este tipo de técnicas. Hay mil y una variantes de la PCR, pero no os torturo más.






No hay comentarios:
Publicar un comentario