Anteriormente en FoxP2... Previously on FoxP2...
Descubierto en pulmón [1], pero rápidamente asociado al lenguaje [2], el gen FoxP2 empieza a salir a la luz de los neurobiólogos [3], y ornitólogos [4].
El tema de los pajaricos seguiría dando de sí. Un año después (2005) aparecía este trabajo (aquí) en el que se secuenciaba el gen FoxP2 de diferentes aves cantoras (el artículo no es de pago, así que podéis acceder a él y ver la lista de las especies analizadas) para luego comparar su secuencia entre sí y con otros vertebrados, como los humanos. Los resultados indicaban que no había variaciones importantes entre las aves, pero sí entre éstas y los humanos (lógico, estamos más separados evolutivamente). Las mutaciones que aparecen en el FoxP2 humano no aparecen tampoco ni en delfines, ballenas o murciélagos. Dicho de otra manera, la ristra de letras del gen de los humanos es "único"... ¡ah! nuestro antropocentrismo respira aliviado. ¿Tiene mucha importancia este último dato? No. Las diferencias en las secuencias de los genes no son raras y aumentan con la distancia filogenética de dos especies. Es decir, dos especies que se separaron evolutivamente hace mucho tiempo tendrán las secuencias de genes más distintas que las especies que hace menos tiempo que divergieron. Por ejemplo, los genes de rata y ratón son mucho más parecidos en su secuencia que los de rata y gusano. Se pueden construir "árboles" de los genes basándonos en las similitudes de sus secuencias de nucleótidos. Aquí tenéis el de la familia FoxP (al que he llegado por este camino: Pubmed gene, homologene, orthology, y treefam, recursos todos ellos gratuitos).
En este árbol se representan los genes y secuencias susceptibles de ser genes de esta familia. Como cualquier árbol genealógico, contra más cerca están dos genes, más emparentados están; más tiempo hace que sus caminos evolutivos divergieron de los de sus "tatarabuelos". Lo primero en lo que nos tenemos que fijar es en los grandes grupos que aparecen (marcados con colores). En azul vemos los FoxP3, los "primos lejanos" del resto (fijaos en que su línea principal, la del "72", sería hija de la del "53" que nos queda a la izquierda, y por tanto, sólo tendría una "hermana", la "100" de la que salen todos los otros grupos coloreados). Estos otros grupos son FoxP4 en verde, FoxP1 en rosa y nuestro FoxP2 en amarillo. Fuera de la coloración encontramos genes parecidos (homólogos) a los FoxP de moscas (DRO) y gusanos (CAE y SCH). Centrémonos en FoxP2.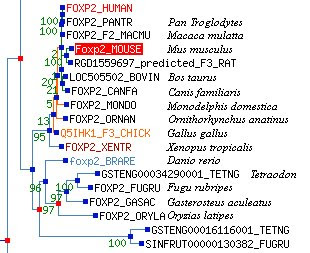
Como hemos dicho, a más alejadas se encuentran dos especies, más diferencias presentan sus genes homólogos. En este sentido los árboles derivados de la comparación de la secuencia de los genes se pueden utilizar como árboles genealógicos de las especies. Esto suponiendo que se hayan acumulado las mutaciones de manera constante en todas las líneas evolutivas. Si, como parece desprenderse del artículo que ha desencadenado toda esta reflexión, existe alguna línea "privilegiada" que acumula más "mutaciones" que otras que justifiquen la "especialidad" de esa especie, el gen de esa especie no se encontrará entre los genes homólogos de las especies más cercanas, sino que se encontrará "a parte" de éstos. Pero, oh tragedia, no es así.
En el zoom podéis ver cómo el gen FoxP2 de humanos está bien cerquita de los chimpancés (Pan) y Macacos. Este "trío" de homólogos está muy emparentado con los FoxP2 del resto de los mamíferos, ratones (Mus), ratas, toros (Bos), perros, delfines, y, a cierta distancia, ornitorrincos. Relacionado con este "pack" encontramos el FoxP2 del gallo. Y a más distancia el de una rana (Xenopus). Este grupo de los "vertebrados terrestres" está emparentado con el resto de genes FoxP2 que corresponden a los de los peces (Danio, Tetraodon, Fugu, Gasterosteus, y Oryzias). Queda claro que el FoxP2 de humanos está donde tiene que estar por secuencia, y que no tiene nada "más especial" en cuanto a su ristra de nucleótidos que el del resto de organismos.
En la siguiente entrega pasaremos al 2005, la perspectiva humana.
30/12/08
FoxP2. V. Pájaros y árboles.
Publicado por
Salva
en
19:46
0
comentarios
![]()
Etiquetas: Biología molecular, Evolución, Genética, Neurobiología, Series divulgativas
29/12/08
Alibri: ¡Qué fácil es tener una buena librería!
Hoy estoy contento. Igual que hace unos días me quejaba del desastroso método de clasificación de la FNAC (que, por vuestros comentarios, resultó no ser un hecho puntual), me toca ahora comentar la profesionalidad de la librería Alibri.
Educados, amables, informados y eficientes. ¿Qué más se puede pedir? Pues que los libros estén bien cuidades, clasificados y expuestos. Qué fácil, ¿verdad?. Lo mejor de esta librería es que en todo momento tienes la sensación de que realmente es muy fácil tener una librería adecuada para los lectores... cuando los encargados son lectores, por supuesto (y por su puesto, tenía que hacer el chascarrillo).
Y no hablo tan solo de ciencia; los otros libros que íbamos a buscar (uno de teoría musical y otro de chino... mejor no preguntéis) también eran fácilmente localizables. ¡Ah! Después de la pesadilla de los "grandes almacenes", una librería como esta siempre te anima el día. No dejéis de visitarla si tenéis la ocasión...
Alibri
Llibreria Universitària Internacional des de 1925.
Carrer Balmes 26.
Barcelona.
Publicado por
Salva
en
15:41
0
comentarios
![]()
Etiquetas: Opinión
28/12/08
FoxP2. IV. Pajarito, ¿cómo estás?
Anteriormente en FoxP2. Previously in FoxP2...
Tras su descubrimiento en pulmón de ratón [1], FoxP2 se ve implicado en la aparición del lenguaje humano [2]. Los estudios sobre este gen se suceden [3]...
2004-2005: Blackbird singing in the dead of night
Entramos en 2004 con un artículo en el que se analiza la expresión de los dos genes (FoxP1 y FoxP2) en pájaros cantores (el diamante mandarín, Taeniopygia guttata). Recordemos que FoxP2 se encuentra en todos los vertebrados, y los diamantes mandarines no son una excepción. Estos pájaros "modulan" su voz a lo largo de su vida como un sistema de comunicación, como nosotros. Por tanto, parece que los resultados que se deriven del estudio de estos pájaros serán extrapolables a humanos. Veamos cuáles fueron estos resultados.
Este artículo demuestra que FoxP2 y FoxP1 se expresan en el cuerpo estriado también de los diamantes mandarines, incluyendo las secciones de este cuerpo encargadas de la integración de estímulos sensomotrices y del control de movimientos precisos y coordinados (los cuales intervienen tanto en el habla como en el "canto"). Apuntan, además, al probable papel de FoxP1 en alguno desórdenes lingüísticos, puesto que se expresa en los mismos lugares del cerebro de estas aves.
En junio de ese año la directora del centro donde se produjo este estudio, publicaba ya un review... ninguno de los otros firmantes del primer artículo aparecía en este último. Por cierto, ¿el título del artículo de junio? Componentes genéticos del aprendizaje vocal. (un par de años después el mismo grupo de investigación comprobaría que la cantidad de proteína FoxP2 disminuye en el cerebro de los pájaros machos que cantan para ellos mismos, pero no en los que cantan para las hembras. Si es que la soledad es muy mala. Bromas a parte, estos resultados demostraron que la expresión de FoxP2 no es fija, sino que depende de factores externos).
En la entrada siguiente seguiremos dando el cante y os presentaremos el árbol de familia de los genes FoxP.
Publicado por
Salva
en
18:17
0
comentarios
![]()
Etiquetas: Biología molecular, Evolución, Genética, Neurobiología, Series divulgativas
25/12/08
FoxP2. III. Evolución, cromosoma y cerebro.
En la entrada anterior. Previously on FoxP2... .
Un gen descubierto en pulmón se convierte en el principal culpable de una deficiencia en el habla. Se prepara el gran salto para ser reconocido como... "el gen del habla". Pero, ¿es esto cierto? ¿qué otros secretos y polémicas encierra este gen?. Continuamos con nuestra historia.
Si FoxP2 tenía que ser un gen clave en nuestra evolución, tenía que serlo. En diciembre de 2002 apareció un artículo en el que se analizaba la tasa de cambios (mutaciones) en la secuencia de diferentes proteínas de humanos, chimpancés y ratones, con el argumento de que "los genes responsables del fenotipo humano deben haberse encontrado bajo presiones selectivas alteradas durante la evolución humana y, por tanto, mostrarán cambios en la tasa de sustitución a nivel de su secuencia proteica". Vamos, que los genes que "nos hacen humanos" (fenotipo) presentarán más cambios que los que no importaron tanto en nuestra "carrera" homínida. De sus análisis emergen dos genes, PRM2, implicado en la generación de espermatozoides, y que ellos relacionan con la presión selectiva sexual (?), y, como no, FoxP2. Comparándolo con otros mamíferos, el FoxP2 de los humanos era el más diferente (sic). Con estos resultados se afirma que FoxP2 "puede haber jugado un papel en el origen del habla humana". Os recomiendo que os leáis el abstract (la introducción-presentación del artículo), vale la pena. Si además queréis leéroslo entero, podéis, pues el artículo no es de pago.
2003: Cromosomas y cerebros
En enero de 2003 aparece un nuevo review, Descifrando las bases genéticas de los desórdenes del habla y del lenguaje en el que se vuelve a hablar de los diferentes orígenes de estos desórdenes y en el que se señalan otras regiones de los cromosomas 2, 13, 16 y 19, como responsables de algunos de estos desórdenes. A FoxP2 le aparecían compañeros... (estos mismos resultados se pueden ver comentados en este artículo del grupo de investiación colombiano encabezado por D.A. Pineda)
El análisis de FoxP2 iba avanzando y se iba estudiando dónde se expresaba (encontrándose, entre otros, en el cerebro) y qué especies lo presentaban. FoxP2 existe en todos los vertebrados en los que se ha buscado. Incluído el pez cebra. En junio de 2003 ya se hablaba de estos descubrimiento, pero esto no impedía a Marcus y Fischer defender el papel de FoxP2 en el lenguaje ya que este gen "puede, sin ser específico de cerebro (recordad que también se expresa en pulmones) o de nuestra especie, proporcionar un incomparable punto de entrada en el conocimiento de las cascadas genéticas y los caminos neuronales que contribuyen a nuestra capacidad para el habla y el lenguaje". (Dos años después aparecerá un artículo de revisión de resultados en el que se comentarán los últimos avances en este sentido).
En julio de 2003 se precisó un poco más en el lugar de expresión de FoxP2 en el cerebro. El cerebro es un órgano extremadamente complejo. Pese a esta complejidad parece que exite cierta especialización de sus regiones. Resulta que FoxP2 (y su "primo-hermano" FoxP1) se expresa en el Cuerpo estriado, una región especializada, según parece, en la producción de movimiento y de otras respuestas, así como de la memoria a largo plazo de secuencias de movimientos asociades a "habilidades" (procedural memory). El habla podría ser una de estas "habilidades", con lo que, según sus autores, los resultados de este artículo refuerzan aún más el papel de FoxP2 en el habla.
En la siguiente entrega, alguien volará sobre el nido del cuco
Publicado por
Salva
en
21:06
0
comentarios
![]()
Etiquetas: Biología molecular, Evolución, Genética, Neurobiología, Series divulgativas
24/12/08
Para nuestros seguidores de RSS
Buenas! Hemos estado haciendo una especie de mantenimiento en Feedburner para poder ver mejor las estadísticas... y puede que haya problemas con las URLs de vuestras subscripciones.
Si es así, intentad volver a suscribiros en "ADSADN a domicilio" de la barra de la derecha. Si incluso así no hay manera de que nos leáis... decídnoslo que me fustigaré por no tocar lo que no domino (¡html...caca!) y luego intentaré arreglarlo.
Molesten las disculpas.
Publicado por
Salva
en
10:25
1 comentarios
![]()
Etiquetas: General
23/12/08
Visitas del último año
Bueno. Hoy entrada "recapitulatoria".
Llevamos ya un poco más de un año en antena y me apetece mostraros la evolución de las visitas en este tiempo que hemos podido seguir gracias a Statcounter.
Aquí podéis ver la evolución de las visitas de la versión en catalán, con un total de 7.856 visitas des del 23 de diciembre del año pasado. 
Y aquí las de la versión en castellano (24.754 visitas).
Muy diferentes, ¿verdad? Creemos que es debido a que la versión en catalán se dio a conocer antes que la de castellano, la cual ha tardado en arrancar, pero cuando lo ha hecho nos ha sorprendido incluso a nosotros mismos... Y todo gracias a que muchísima gente del Cono sur, México y New York (principalmente) han empezado a visitarnos (¡muchísimas gracias!)
A lo largo de estos meses ha habido de promedio un 70% de las "visitas" que han durado menos de 30 segundos (para qué os voy a engañar). O es gente que lee muy rápido, o se trata de buscadores que barren las páginas web. Con lo que el total de personas que realmente nos habrán leído es de 2.356,8 personas en catalán, y 7.426,2 personas en castellano... Un total de 9783 personas. Sé que no es mucho, pero ¡os podéis imaginar la ilusión que nos hace un número casi de cinco cifras!
Teniendo en cuenta que hemos publicado 149 entradas en este periodo de tiempo, ha habido una media de 65,6 personas que se han leído cada una de nuestras entradas. ¡Muchas, muchísimas gracias! Esperamos volver a veros de vuelta más de una vez... Y para cualquier sugerencia, no dudéis en escribirnos o dejar algún comentario.
Nos vemos.
Publicado por
Salva
en
22:28
0
comentarios
![]()
Etiquetas: General
21/12/08
The unfolding of language. La evolución del lenguaje
 Será quizás por la cercanía de las fiestas, por las reuniones familiares, por las cenas de empresa, o por la desinhibició propia del vino y el cava que nos hemos puesto tan parlanchines. Sea como sea, llevamos ya un par de entradas tratando el tema del habla desde su vertiente más "molecular" (y alguna anterior desde otros puntos de vista: [1], [2], [3], y [4]), y, como preveo que irá para largo (me sigue pareciendo un tema interesantísimo), nos permitiréis hacer un alto en el camino y recomendaros un libro espectacular muy relacionado con este tema: The unfolding of language. The evolution of mankind's greatest invention., de Guy Deutscher.(¡Gracias, Oriol!)
Será quizás por la cercanía de las fiestas, por las reuniones familiares, por las cenas de empresa, o por la desinhibició propia del vino y el cava que nos hemos puesto tan parlanchines. Sea como sea, llevamos ya un par de entradas tratando el tema del habla desde su vertiente más "molecular" (y alguna anterior desde otros puntos de vista: [1], [2], [3], y [4]), y, como preveo que irá para largo (me sigue pareciendo un tema interesantísimo), nos permitiréis hacer un alto en el camino y recomendaros un libro espectacular muy relacionado con este tema: The unfolding of language. The evolution of mankind's greatest invention., de Guy Deutscher.(¡Gracias, Oriol!)
¿Pueden los idiomas modernos, con su "coherencia interna" y su "complejidad" haber surgido "naturalmente" desde un pre-lenguaje con unos elementos concretos y limitados? ¿No es más lógico pensar que esta "plenitud" de los idiomas es resultado del "diseño" de unos cuantos lingüistas, como los idiomas de El Señor de los Anillos? ¿No os suenan estos argumentos? ¿Aparecerán también grupos de presión linguocreacionistas? ¿Se escandalizarán las familias reales por estas ideas? ¿Ofrecerán dinero a quien les traiga pruebas de esta "evolución"? Porque las hay a miles... Pero hemos venido aquí a hablar del libro.
Llevo un mes disfrutando como un niño cada noche leyéndomelo, tanto por la calidad didáctica de su escritura (un ejemplo brillante de una trabajadísima ordenación de ideas, una exposición transparente, y la dosis justa de humor y de propuestas arriesgada), como por su contenido. El autor nos lleva de la mano por los diferentes capítulos del libro en los que nos va presentando los principales procesos que han ido modelando los idiomas. Las ideas son buenas, la exposición mejor, y su lectura asegura horas de divulgación de alto nivel, amena, inspiradora y enriquecedora... ¿Qué más se le puede pedir? Nada de nada. Un libro redondo.
Publicado por
Salva
en
12:32
0
comentarios
![]()
Etiquetas: Antropología, Evolución, Lecturas recomendadas
18/12/08
FoxP2. II. 2001 Odisea en el lexinoma
En la entrada anterior... Previously on FoxP2...
2001-2002: El monolito del lenguaje (Reprise)
Empecemos por el principio. FoxP2 aparece por primera vez mencionado en unartículo del año 2001 (es un gen "joven"). ¿Fue identificado en el cerebro? ¿Cantaba? No. El artículo de Weigo Shu y colegas identifica dos nuevos genes, FoxP1 y FoxP2, en pulmón de ratón y actuando como represores. Es decir, impidiendo que determinados genes se expresen. ¿Pulmón? ¿Represor? ¿Existirá entonces un "gen del silencio" inhibido por FoxP2?
El artículo anterior apareció en julio del 2001. En octubre se publica la primera relación entre este gen y el habla. El equipo de Oxford de Anthony Monaco (recordad que el jefe siempre firma el último) llevaba tiempo estudiando personas con déficit en el habla y había encontrado una familia (familia KE) en la que este defecto se heredaba como si se tratase de una enfermedad genética "mendeliana" (que depende de un solo gen). Habían llegado incluso a identificar en qué región del cromosoma 7 humano se encontraba el problema. Y en esa región estaba FoxP2. Los individuos que tenían afectada la región del cromosoma 7 donde se halla FoxP2 presentaban dificultades en el habla. ¡Bingo! Si no tienes el gen y tienes dificultades al hablar, esto quiere decir que el gen es necesario para esta función. Listo. Tan importante descubrimiento mereció comentarios tanto en el propio Nature, como en su "rival" Science, en el que, por cierto, se refieren a nuestro protagonista como el "primer" gen del habla.
Diferentes artículos aparecidos posteriormente buscaron este gen en otros pacientes con déficits en el habla y no encontraron relación con FoxP2. Dicho de otra manera, a pesar de tener "bien" el gen FoxP2, los pacientes presentaban carencias a nivel del lenguaje (como el caso de los 270 niños de 4 años analizados en este artículo). Queda claro, pues, que FoxP2 no es el único gen del habla, porque hay gente con problemas, a pesar de que su FoxP2 es tan bueno como el de cualquier otro.
Estos resultados no impidieron que se desatara la euforia y que incluso Science se atreviera a calificarlo como el "gen del habla" en este review del 2002 (los review son artículos en los que se intenta poner todo lo que se conoce sobre un tema en concreto; contienen muchas referencias y normalmente ningún experimento). Rizando el rizo, otros de estos reviews lo incluían entre "los genes que nos hacen humanos".
Por otro lado se publicaron otros trabajos que pretendían sembrar cordura en este terreno. Es el caso del artículo de Diane Newbury y el propio Anthony Monaco con el que pretenden analizar el impacto, un año después del descubrimiento de la relación de FoxP2 con el habla, siendo que ésta se había demostrado solamente en una familia.
En la próxima entrega cambiaremos por fin de año (2002) y veremos cómo FoxP2 se convierte en una nueva diana evolutiva y neurobiológica.
Publicado por
Salva
en
22:30
0
comentarios
![]()
Etiquetas: Biología molecular, Evolución, Genética, Neurobiología, Series divulgativas
16/12/08
FoxP2. I. Introducción. El gen ¿parlante?
 Hace un poco más de un mes, Quim nos habló, en su entrada El gen de los ojos verdes, sobre la confusión que existe entre la función de los genes y lo que éstos "hacen". La entrada incidía sobre el error común que representa el creer que existe un gen para el color de los ojos, para la hemofilia o para tener unas proporciones helénicas perfectas. Tampoco existe el "gen del habla", por mucho que hace unos años se comentase en más de un "noticiario". Este gen en cuestión es el FoxP2. ¿Es realmente el gen del habla? ¿Es el único?
Hace un poco más de un mes, Quim nos habló, en su entrada El gen de los ojos verdes, sobre la confusión que existe entre la función de los genes y lo que éstos "hacen". La entrada incidía sobre el error común que representa el creer que existe un gen para el color de los ojos, para la hemofilia o para tener unas proporciones helénicas perfectas. Tampoco existe el "gen del habla", por mucho que hace unos años se comentase en más de un "noticiario". Este gen en cuestión es el FoxP2. ¿Es realmente el gen del habla? ¿Es el único?
Por partes. Los genes codifican para proteínas. Lo que quiere decir que a partir de ellos se construyen determinadas proteínas. Y nada más. ¿Cómo puede el gen FoxP2 ser el gen del habla... ?¿Qué hace? ¿Una proteína que suena?
Las proteínas pueden tener diferentes funciones. Algunas son meras estructuras sobre las que se sostiene y vive la célula. Otras son capaces de "hacer cosas" (las famosas enzimas). Y en otro grupo encontramos las que son capaces de unirse al DNA y controlar la expresión de diversos genes, es decir, su transcripción... FoxP2 es uno de estos factores de transcripción (comentados ya Back to the future, Origen genético y epigenético de la magia, o ¿Quién corrompe a los linfocitos?). O sea que su función es controlar la expresión de otros genes, que a su vez sirven de plano para la construcción de nuevas proteínas, etc...
Veamos qué conocemos hasta el momento de FoxP2 y veremos a qué conclusión llegamos.
2001-2002: El monolito del lenguaje
Empecemos por el principio. FoxP2 aparece por primera vez mencionado en un artículo del año 2001 (es un gen "joven"). ¿Fue identificado en el cerebro? ¿Cantaba? No. El artículo de Weigo Shu -y colegas- identifica dos nuevos genes, FoxP1 y FoxP2, en pulmón de ratón y actuando como represores. Es decir, impidiendo que determinados genes se expresen. ¿Pulmón? ¿Represor? ¿Existirá entonces un "gen del silencio" inhibido por FoxP2?
En las siguientes entregas de FoxP2, el gen parlante, comentaremos más artículos que se han ido publicando desde su descubrimiento y veremos por qué se ha ganado la fama que tiene.
Publicado por
Salva
en
8:00
0
comentarios
![]()
Etiquetas: Biología molecular, Evolución, Genética, Neurobiología, Series divulgativas
14/12/08
Lab Basics (9): Fábricas de proteínas
 Todas las técnicas de laboratorio que hemos estado explicando en las entradas anteriores del blog consisten en maneras de aislar, detectar, purificar, analizar y modificar el material genético según nuestros propósitos. A menudo, el fin mismo de nuestros experimentos es ese, pero a menudo todo el trabajo anterior tiene como finalidad que ese material genético se traduzca en proteínas. Si queremos seguir un proceso metabólico o una función dentro de la célula, necesitamos la proteína, no la receta de cómo hacerla. La célula es la fábrica en la que se producirá nuestra proteína de interés, y ya vimos cómo tener nuestro huerto de células particular.
Todas las técnicas de laboratorio que hemos estado explicando en las entradas anteriores del blog consisten en maneras de aislar, detectar, purificar, analizar y modificar el material genético según nuestros propósitos. A menudo, el fin mismo de nuestros experimentos es ese, pero a menudo todo el trabajo anterior tiene como finalidad que ese material genético se traduzca en proteínas. Si queremos seguir un proceso metabólico o una función dentro de la célula, necesitamos la proteína, no la receta de cómo hacerla. La célula es la fábrica en la que se producirá nuestra proteína de interés, y ya vimos cómo tener nuestro huerto de células particular.
Introducir material genético (por ejemplo, nuestro gen clonado en un vector bacteriano) dentro de la célula no es sencillo: los sistemas celulares son impermeables al paso de material genético foráneo como sistema de protección ante los virus. El gran escollo a salvar es la membrana citoplasmática (y en eucariotas, la nuclear).
Existen multitud de métodos para forzar a la célula a aceptar material genético extraño. Un vector con un gen clonado puede funcionar de forma autónoma (contiene sus propios orígenes de replicación) o integrarse en algún lugar del genoma. A menudo el experimento no requiere nada más que expresar que la proteína de interés se traduzca (24 o 48 horas es más que suficiente): del extracto de células se purifican la proteínas. Un material genético que no se haya integrado en el propio genoma de la célula, se perderá en las sucesivas divisiones de la célula.
A veces interesa seleccionar sólo las células que hayan incorporado el vector de expresión de forma permanente; para ello se tratan con concentraciones adecuadas del antibiótico al cual el vector de clonaje es resistente. Morirán todas las células que no hayan incorporado el vector en su genoma de forma estable. Pero ojo, la integración en el genoma del vector no es dirigida y por lo tanto podemos estar dañando la célula. Para que nuestro gen foráneo se integre de forma permanente en un lugar concreto del genoma que a nosotros nos interesa se necesita una odisea de técnicas de ingeniería genética que no detallaremos.
Una manera brava de permeabilizar las membranas lipídicas es agujerearlas con detergentes suaves o un choque osmótico, pero hay alto riesgo de romperlas. Otro sistema es envolver el material a introducir en un agregado que la célula fagocite de forma natural. Si el vector a introducir es grande, se pueden electrocutar las células durante pocos segundos para agujerear sus membranas (electroporación). Las células despolarizan sus membranas y se forman pequeños orificios por los que penetran las moléculas (proteínas, ADN...). Incluso se puede disparar a las células con unas pistolas especiales en las cuales el material genético se une a una nanomolécula (suele ser oro), a forma de balín que llegará al núcleo celular.
Todos estos procedimientos causan gran mortaldad en las células.
Si queremos que nuestras fábricas de proteínas sean células bacterianas (no siempre procesan igual de bien las proteínas de mamífero que las células eucariotas pero es fácil tener volúmenes industriales de cultivos), también se pueden agujerear las membranas por electroporación o choque térmico, aunque son cepas de bacterias tratadas previamente con productos químicos a fin de que su membrana sea más susceptible a formar agujeros (células competentes, que se llaman).
Para algunas aplicaciones concretas, se microinyecta en la célula lo que se desea introducir (es la típica imagen del oocito al que se le pincha un espermatozoide). Es extremadamente delicado, difícil y costoso y requiere una micropipeta que se controla con la ayuda de un micromanipulador bajo un microscopio. La mortaldad de las células también es muy alta.
Lo más usado hoy en día es el sistema de lipofección. Consiste en envolver el material genético a introducir en una gabardina de moléculas lipídicas con carga positiva que tienen afinidad con las membranas biológicas, de manera que se arrimen a la membrana y depositen su carga dentro de la célula. Los compuestos de lipofección son caros (cifras de tres digitos) y hay que ensayar las condiciones para cada tipo celular, pero el protocolo en sí es sencillo y rápido.
Otra manera tan eficiente o más que la lipofección pero también más peligrosa para el investigador es utilizar esas jeringuillas naturales que son los virus: virus modificados genéticamente que contengan en su genoma nuestro gen de interés, obraran sus malas artes para que ese material acabe dentro de la célula. Es un proceso mas largo y complejo pero si las células lo permiten, tiene una eficiencia nada desdeñable. El trabajo en laboratorio con virus modificados requiere medidas extras de seguridad cuando se trata de virus que pueden penetrar en células de mamífero (adenovirus por ejemplo).
Forzar a una célula a fabricar ingentes cantidades de nuestra proteína de interés supone un estrés para la fábrica (cual empresa turronera en diciembre), que tiene que destinar muchos recursos a una proteína que a menudo le es extraña en vez de dedicarse a otros menesteres. Pese a que pueda parecer un sistema artefactual, nos tenemos que conformar porque a veces es la única manera de poder estudiar en sistemas vivos algunos procesos.
Sirvan estos breves comentarios para ilustrar la complejidad de que un gen entre en una célula así como así: no sólo tiene que traspasar unas membrana impermeables si no que además, o se integra con ínfima probabilidad en el genoma o más vale que forme parte de alguna estructura biológica que garantice su perpetuidad y transcripción (cromosoma bacteriano, virus). Pequeñas menudencias que los gurús que nos advierten de los peligros de ingerir alimentos que contengan genes malignos de otras especies no parecen tener en cuenta. La transferencia de genes horizontal es común en bacterias y según la teoría endosimbiótica ha ocurrido en eucariotas, pero no pasa de ahí.
Publicado por
Elena Garrido
en
12:04
1 comentarios
![]()
Etiquetas: Series divulgativas
12/12/08
El (ADS)L: entradas interes(ADN)tes, 12-12-08
Esta semana hemos leído y os recomendamos:
- Ositos de peluche en el espacio. Tremenda historia y acongojantes fotos sobre un experimento realizado por alumnos de primaria. Ovación para el profesor. Seguro que en esa clase hay más de un futuro científico gracias a esta iniciativa.
- Un día cualquiera: fuera de la cama, dormilón, en Genciencia. De cómo se nos alteran las fases del sueño con la edad. Y yo añadiría... ¡y con el alcohol! (la entrada anterior del blog también vale la pena, Un día cualquiera: suena el despertador, y sobre el tema de los ritmos circadianos, recomedaros esta entrada de la revista Eureka).
Os recomendamos además:
- Y hablando de alcohol freshquito, aquí tenéis esta etrada: Enzims implicats en l'elaboració de la cervesa (en catalán), en The Industrial Enzymologist.
- Desacobla't bé, fill meu, que fa fred(en catalán), en Abulafia. Viaje en forma de entrada entre mamuts, humanos, neandertales, mitocondrias, enzimas, ATP, protones... para acabar mirando de cerca nuestras pisadas en la luna. ¿podrían estar los neandertales demasiado adaptados al frío?
- Llibres i més llibres, en Històries de la ciència (en catalán). Ciencia y libros. Irresistible.
Publicado por
Salva
en
10:00
0
comentarios
![]()
Etiquetas: Ciencia en los medios
10/12/08
La ignorante manera de clasificar libros de ciencia
19:30. Diagonal de Barcelona. FNAC.
No puedo evitarlo. Cada vez que paso por cualquier librería la "deformación profesional" me lleva a buscar la sección de "ciencias" para comprobar tanto su contenido como la extensión que se le dedica. En la FNAC se llama "Divulgación científica", está situada al lado de Ciencias sociales (con especial mención a "Autoayuda") y ocupa dos columnas. Cada fila de libros tiene más libros detrás, prácticamente inaccesibles. Esta vez no miro por encima los títulos que exponen.
Por curiosidad, he decidido buscar si tienen las últimas novedades de las que os he hablado: Conviviendo con transgénicos y El genoma fluido. El primero no lo encuentro. Qué raro. Voy a preguntar... Sí, sí lo tienen... en la sección de Salud. ¡¿Qué?!
Bueno, no nos pongamos nerviosos. Alguna explicación tiene que haber. Tranquilo. Respira. No puede ser. Es un error. Quizás salud es una sección dedicada a las "biociencias" y la medicina. Quizás... No.
Ahí está. En la última fila de la tercera columna de Salud. Al lado de Nutrición de la A a la Z y, os lo juro, Adelgazar follando. Ni rastro de biología. Ni rastro tampoco de libros sobre transgénicos.
Volvamos al mostrador para avisar. Al fin y al cabo ya me había encontrado con algo parecido con el libro de Lynn Margulis y Dorion Sagan, ¿Qué es el sexo?, un libro sobre la aparición de la reproducción sexual en la evolución y que, evidentemente estaba entre los libros de sexualidad humana. Avisé. Y se solucionó. Pero esta vez no es tan fácil. La dependienta, muy amable y simpática todo sea dicho, nos dice que no es el primer caso de ordenación absurda que reciben, ya que ellos se dedican a ordenar los libros según un código que les viene impuesto desde Madrid... Si es así. Gracias. No, gracias a vosotros, y siento no poder hacer nada. No, mujer, no. En fin. Flaco favor se le hace al libro colocándolo donde está. ¿Quién será el ignorante que habrá decidido colocarlo en "Salud"? ¿Cómo ha tomado esta decisión?
En fin. Así se entiende la ciencia. Qué pena.
Publicado por
Salva
en
22:04
3
comentarios
![]()
Etiquetas: Ciencia en los medios
7/12/08
Paranoia Protbook: ¿podremos desentrañar el funcionamiento de las células?
El ser humano está "condenado" a sentir verdadera fascinación por los conceptos que no comprende. Esta fascinación es la madre de la ciencia y de la imaginación. Desde hace muchos años me siento poderosamente atraído por el concepto de entropía, de lo que deduzco que mucho no lo acabo de entender. La entropía se puede definir de muchas maneras, pero para lo que hoy me interesa, nos quedaremos con que es el "desorden" de las partículas de un sistema. El ejemplo paradigmático es el de una caja con una separación en medio que tiene a un lado gas, y al otro, vacío. Cuando se quita la separación las moléculas del gas ocupan el espacio vacío y se alcanza un estado de equilibrio. En este último estado hay más "desorden" que al principio (las moléculas antes estaban "ordenadas" a un lado de la caja y ahora están dispersas por toda la caja).
La entropía se puede definir de muchas maneras, pero para lo que hoy me interesa, nos quedaremos con que es el "desorden" de las partículas de un sistema. El ejemplo paradigmático es el de una caja con una separación en medio que tiene a un lado gas, y al otro, vacío. Cuando se quita la separación las moléculas del gas ocupan el espacio vacío y se alcanza un estado de equilibrio. En este último estado hay más "desorden" que al principio (las moléculas antes estaban "ordenadas" a un lado de la caja y ahora están dispersas por toda la caja).
Lo que encuentro más fascinante es el paso del sistema pequeño al grande. Me explico. Si en el caso de la caja nos fijamos en una sola de las partículas, aparentemente no pasa absolutamente nada cuando quitamos la separación. Es cuando miramos el conjunto de las partículas cuando emerge la entropía. Hay una barrera de magnitud por debajo de la cual no es medible la entropía. Me encantaría saber dónde está situada esta barrera; o dicho de otra manera, cuántas partículas hay que considerar para que emerja la entropía (¿dos? ¿diez a la 23?)
Esta barrera de magnitud creo que es también aplicable a procesos biológicos. Si miramos una gacela podemos hacernos una idea de cómo funciona individualmente (cómo se mueve, cómo se alimenta, etc)... pero si consideramos su "rebaño" emerge una nueva magnitud: sus relaciones sociales; y si consideramos su entorno, emerge otra, las relaciones ecológicas.
Y lo mismo sirve para escalas menores. Podemos hacer todos los artículos del mundo sobre un tipo de célula aislada de ratón, cultivada en una placa de plástico con suero de ternera y tratada con algún compuesto disuelto en alcohol (todo muy "natural"), pero cuando la consideremos dentro del cuerpo, aparecerá otra magitud de relaciones que harán surgir una nueva medición (una entropía celular si queremos llamarla así). Pensad si no en las neuronas. Podemos entender, más o menos, cóo funciona una neurona. Pero, ¿podremos entender a partir de estos datos cómo se generan los recuerdos? ¿O los pensamientos?
Aún más. Si nos dedicamos a estudiar la relación de una proteína en concreto con otra de su entorno bajo unas condiciones concretas podemos obtener datos y más datos (si es una enzima, por ejemplo, a qué velocidad trabaja, cuándo se satura, con quién interacciona, cuál es la cantidad óptima de sustrato, etc.), pero, de nuevo, cuando la consideremos en conjunto con la miríada de otras proteínas disueltas en el plasma de nuestras células corremos el riesgo de perder la perspectiva de nuevo. Hace ya años que se publican miles de artículos mensuales sobre biología molecular. Estos artículos aportan pequeñas informaciones sobre proteínas concretas. Faltaría empezar a aglutinarlas. Algunos centros ya lo están haciendo, pero es un trabajo ingente. Y aquí viene la paranoia protbookista del título. ¿Y si empezamos a aprovechar las posibilidades de internet para generar redes de proteínas "sociales"? Ya se ha comentado (en otros blogs, como Abulafia y Vallve's Blog y en este mismo) los primeros pasos en este sentido. Pero podríamos ir más allá y montar un proyecto tipo Seti@home. En este proyecto "cedes" parte de la potencia de tu ordenador para "escanear" el cielo en busca de señales de vida extraterrestres. ¿Por qué no aprovechar la idea y hacer una gran red de proteínas "virtuales"? Cada persona podría "escoger" una proteína concreta. Las conexiones se establecerían teniendo en cuenta las interacciones que se dan entre las proteínas. Podríamos, incluso, aprovechar, las plataformas sociales ya existentes tipo Facebook (ya que estamos, nos podréis encontrar ahí). ¿Os parece factible? ¿O es producto del cóctel de fármacos que me he tomado para luchar contra el trancazo que llevo?
Hace ya años que se publican miles de artículos mensuales sobre biología molecular. Estos artículos aportan pequeñas informaciones sobre proteínas concretas. Faltaría empezar a aglutinarlas. Algunos centros ya lo están haciendo, pero es un trabajo ingente. Y aquí viene la paranoia protbookista del título. ¿Y si empezamos a aprovechar las posibilidades de internet para generar redes de proteínas "sociales"? Ya se ha comentado (en otros blogs, como Abulafia y Vallve's Blog y en este mismo) los primeros pasos en este sentido. Pero podríamos ir más allá y montar un proyecto tipo Seti@home. En este proyecto "cedes" parte de la potencia de tu ordenador para "escanear" el cielo en busca de señales de vida extraterrestres. ¿Por qué no aprovechar la idea y hacer una gran red de proteínas "virtuales"? Cada persona podría "escoger" una proteína concreta. Las conexiones se establecerían teniendo en cuenta las interacciones que se dan entre las proteínas. Podríamos, incluso, aprovechar, las plataformas sociales ya existentes tipo Facebook (ya que estamos, nos podréis encontrar ahí). ¿Os parece factible? ¿O es producto del cóctel de fármacos que me he tomado para luchar contra el trancazo que llevo?
Y ya que estamos con las paranoias... si se pudiera implementar este programilla en el Facebook, ¿se cumpliría también la regla de los seis nudos? Esta teoría predice que dos personas cualesquiera de todo el mundo están conectadas por seis pasos intermedios de personas conocidas. ¿Habrá igualmente "sólo" seis pasos intermedios entre dos proteínas cualesquiera? ¿Sirve este dato para algo? Buf. Creo que me ha subido la fiebre.
Images:
1. Modified from Wikimedia commons.
2.Kernigh, Wikimedia commons
3. Namazu-tron, Wikimedia commons
Publicado por
Salva
en
11:28
2
comentarios
![]()
Etiquetas: Biología molecular, Opinión
5/12/08
En el (ADS)L: Entradas interes(ADN)tes, 04-12-08
Esta semana hemos leído y os recomendamos:
- El DNA com magatzem de records(en catalán) en Nòmades. Fantástica entrada que nos explica la nueva hipótesis que trata de explicar el mecanismo molecular de la memoria, así como del experimento realizado para ponerla a prueba. Interesantísima. No os olvidéis de visitarla.
- Y como de recuerdos va la cosa, aquí os dejo otra genial entrada: D'estrés i hormones (en catalán), en Abulafia. Buena, buena. Estrés, gatos, leones, novias, pinzas (auch), pasando por glucocorticoides y la relación entre afinidad y plazo de tiempo (y no hablo de relaciones humanas)
También os recomendamos:
- Dos entradas de Tall$Cute: 10 curiosidades bioquímicas sobre nuestro cuerpo, y ¿Qué le preguntarías a un científico español de renombre?. Creo que los títulos lo dicen todo.
- Los políticos no saben qué es el CO2, en E-ciencia. También el título de la entrada lo deja todo clarito.... ¿cómo van a controlar su emisión?
- LA tinta electrónica, en Ocularis. Comparación entre las pantallas convencionales de tele y ordenatas y las pantallas de "tinta digital"... ¿estamos ante un nuevo fenómeno vídeo Beta?
- En apuntes del camino nos recomiendan un mapa del metro de la ciencia de los más famosos científicos del mundo relacionados. Curioso.
Publicado por
Salva
en
8:00
0
comentarios
![]()
Etiquetas: Ciencia en los medios
4/12/08
Artículos sobre "cultura científica"
Los que lleváis un tiempo siguiéndonos sabréis cómo últimamente hemos ido aumentando las entradas relacionadas con la "cultura científica", qué sabe la población sobre ciencia y la imagen que de ella se da en los medios de comunicación.
En esta pequeña entrada de hoy os quiero ofrecer algunos artículos que he ido "acumulando" sobre el tema, para que saquéis vuestras propias conclusiones:
La mayoría de gente, aunque pase por la escuela, es analfabeta científica. Entrevista de Jesús Rubio a Ramón Núñez, publicada en el Diario de Navarra.
Es injusto que un 'mindundi' gane millones cantando o con el fútbol y un científico no. Entrevista de J.L. Obrador a Manuel Toharia, publicada en 20 minutos.
Los mejores estudiantes ya no buscan su futuro en la universidad. Entrevista de César Coca a Margarita Salas, publicada en Laverdad.es.
Es urgente la separación entre rumores y ciencia en internet. Nota de prensa de EFE referente a una entrevista de la BBC a Tim Berners-Lee, leída en Terra.es.
Publicado por
Salva
en
14:00
2
comentarios
![]()
Etiquetas: Ciencia en los medios
2/12/08
Reparaciones que envejecen... SIRT, YES SIRT!
Aunque no lancemos las campanas al vuelo demasiado rápido, ya que queda por comprobar la vinculación que algunos estudios hacen del papel de SIRT1 en la mejora de la supervivencia de células cancerosas (en su esfuerzo por reparar lo que está alterado...)
No, no me he confundido... no quería decir que "embellecen"... (si no, mirad la foto). ¿No os ha pasado nunca que, al abrir una ventana para intentar echar a una mosca, se os ha colado un par de ellas (además de no conseguir echar a la primera, cosa que en general me pasa a mi...)?
Pues eso parece que le pasa también a ciertos mecanismos de reparación de nuestro ADN cuando intenta reducir los efectos que el paso del tiempo tiene sobre su integridad...
Al menos, eso parece desprenderse de lo que publica el biólogo molecular David Sinclair en la revista Cell, donde estudia los efectos que la movilización de la proteína SIRT1 (conocida como reparadora de daños en el material genético de los mamíferos) sobre otros procesos ligados al envejecimiento.
Digamos, para empezar, que uno de los procesos más ligados al envejecimiento es, precisamente, la degradación del ADN. Imaginad: la "copia master" de vuestro disco preferido (ADN) se ha estado utilizando para hacer millones de copias de trabajo (ARN) durante años y años. Pese a que estas copias de trabajo se hacían para que no se estropease la "master", ¡ay...! con los años la cosa empieza a degenerar.
Afortunadamente, todos los organismos cuentan con "proteínas patrulleras", encargadas de reparar desperfectos cuando se detectan signos de que estos daños han podido producirse. Una de las familias de proteínas reparadoras más célebres la constituyen las sirtuinas. SIRT 1 es una de ellas.
Pero, y aqui empiezan los problemas, ya hace años que se descubrió que Sir2, el equivalente de SIRT1 (de mamíferos) que encontramos en las levaduras, "descansaba" sobre una sección del ADN (cuando no tenía que reparar). En esta sección, Sir2 no permitía la expresión del gen que se encontraba codificado (de hecho, Sir2 son las siglas inglesas para "regulador de la información silenciosa"). En cambio, cuando un daño en el ADN mobilizaba la proteína Sir2, la sección ocupada por la proteína en reposo quedaba libre, y la expresión del gen codificado era posible. Lo más impactante es que este gen provocaba la esterilidad de la levadura (una típica característica de senescencia en hongos).
Conclusión: queriendo detener el envejecimiento provocado por las rupturas del ADN, la proteína Sir2 permite la expresión de un gen que provoca envejecimiento... maldita la gracia, que decía mi abuela...
Lo que Sinclair y sus colegas de la Harvard Medical School se han preguntado es "¿hay algun tipo de proceso similar en el equivalente de Sir2 en mamíferos (SIRT1)?"
¿Y qué han hecho? Para empezar, han echado un vistazo a dónde se localiza SIRT1 cuando no está ocupada reparando daños, y han visto que se encuentra enganchada a una región de ADN que no se transcribe, cosa que sugiere el mismo rol "silenciador" que Sir2 tiene en las levaduras.
A continuación, provocaron envejecimiento prematuro con la exposición a peróxido de hidrógeno (ya sabéis que la oxidación es uno de los peligros más grandes que afrontan nuestras células, y se cree que constituye el principal factor de envejecimiento celular) Y se comprobó que más del 90% de la proteína SIRT1 abandonaba su lugar original para dirigirse a las rupturas causadas en el ADN por la oxidación.
Y ahora, el aunténtico objetivo de este estudio: ¿Qué pasaba con las regiones antes ocupadas por SIRT1? Pues los estudios posteriores parecen indicar que los genes en principio silenciados por SIRT1 pasan a expresarse, provocando expresiones anómalas que podrían encontrase en la base de malos funcionamientos y errores propios del envejecimiento celular.
Si esta cuadratura del círculo biológica se confirma, podríamos decir que nos encontramos ante un "callejón sin salida" de la vida: los esfuerzos de la célula para controlar su envejecimiento resultan incluso en su aceleración... Eso sí, tal y como afirma el propio Sinclair, el estudio de SIRT1 puede conducir a mejoras en nuestra comprensión sobre el envejecimiento: no en vano, en su experimento, los ratones a quien se dio un suplemento externo de SIRT1 vivieron más de 25 días más que el grupo control, después de haber estado expuestos a los daños por oxidación...
Publicado por
Quim ADSADN
en
12:18
2
comentarios
![]()
Etiquetas: Actualidad, Genética
29/11/08
Del origen de los pelos ¡Eso pareces tú! ¡Un reptil peludo!
 La entrada de hoy es peliaguda. En el último número del PNAS ha aparecido este artículo relacionado con el "origen" de los pelos de los mamíferos.
La entrada de hoy es peliaguda. En el último número del PNAS ha aparecido este artículo relacionado con el "origen" de los pelos de los mamíferos.
Los mamíferos son, actualmente, los únicos animales con pelo que existen. Los pelos representan una excelente herramienta adaptativa (aunque la gente ahora luche por quitárselos). La "pelambrera" actúa como un aislante térmico, dificulta o ralentiza el intercambio de calor con el medio. Nos mantiene calentitos cuando hace frío.
¿De dónde viene el pelo? Si todos los mamíferos tienen pelo, el antepasado común de éstos también debería tenerlo... y este antepasado fue un reptil. Hace unos 275 millones de años se diferenció un grupo de reptiles, llamados Terápsidos. Éstos se adaptaron a muchos ambientes distintos en lo que se conoce como radiación evolutiva. Muchas especies de terápsidos empezaron a crecer y multiplicarse, constituyéndose en el orden predominante durante unos cuantos millones de años, hasta finales del pérmico, hace unos 250 millones de años. En esta época tuvo lugar la gran extinción del pérmico-triásico. Aunque no es la más famosa (no la confundáis con la de los dinosaurios), ésta es, sin duda, la mayor extinción que se ha dado en el planeta tierra. Más del 90% de las especies marinas y un 70% de las terrestres fueron borradas del mapa. Empezó un nuevo orden en el que fueron unos nuevos reptiles los que se adaptarían a la inmensa mayoría de nichos ecológicos. Hablamos de los dinosaurios.
En esta época tuvo lugar la gran extinción del pérmico-triásico. Aunque no es la más famosa (no la confundáis con la de los dinosaurios), ésta es, sin duda, la mayor extinción que se ha dado en el planeta tierra. Más del 90% de las especies marinas y un 70% de las terrestres fueron borradas del mapa. Empezó un nuevo orden en el que fueron unos nuevos reptiles los que se adaptarían a la inmensa mayoría de nichos ecológicos. Hablamos de los dinosaurios.
Los terápsidos no se extinguieron del todo, aunque ya no volverían a tener el peso en cuanto a número de especies y extensión que habían tenido. Relegados a unos nichos concretos fueron sucediéndose las adaptaciones, y por tanto, las especies, géneros, familias y órdenes. De entre los terápsidos supervivientes cabe destacar el orden de los Eutheriodonta, en el que encontramos el suborden de los Cynodontia. Los cinodontes son los antepasados directos de los mamíferos y se sospecha que ya eran peludos y de sangre caliente. Así que el pelo surgió en algún momento de la historia evolutiva de los terápsidos, antes (un poco o mucho) de los cinodontes. Actualmente no queda ningún superviviente de los reptiles cinodontes (exceptuándonos a los mamíferos, claro). ¿Cómo podemos mirar, entonces, de dónde vienen los pelos? Pues en el DNA. El DNA de cada especie es como una lengua de las que se habla actualmente. La etimología compara cómo son las palabras, cómo se conjugan los verbos, cómo se construyen las frases, y va "tirando" hacia atrás viendo que algunas lenguas actuales comparten una lengua común más reciente que otra. El castellano y el italiano comparten el latín; éstos comparten con el inglés la raiz indo-europea; etc (sobre este tema, recomendaros el libro Unfolding the Language, de Guy Deutscher, un excelente libro del que, seguro, os hablaré cuando me lo acabe).
Actualmente no queda ningún superviviente de los reptiles cinodontes (exceptuándonos a los mamíferos, claro). ¿Cómo podemos mirar, entonces, de dónde vienen los pelos? Pues en el DNA. El DNA de cada especie es como una lengua de las que se habla actualmente. La etimología compara cómo son las palabras, cómo se conjugan los verbos, cómo se construyen las frases, y va "tirando" hacia atrás viendo que algunas lenguas actuales comparten una lengua común más reciente que otra. El castellano y el italiano comparten el latín; éstos comparten con el inglés la raiz indo-europea; etc (sobre este tema, recomendaros el libro Unfolding the Language, de Guy Deutscher, un excelente libro del que, seguro, os hablaré cuando me lo acabe).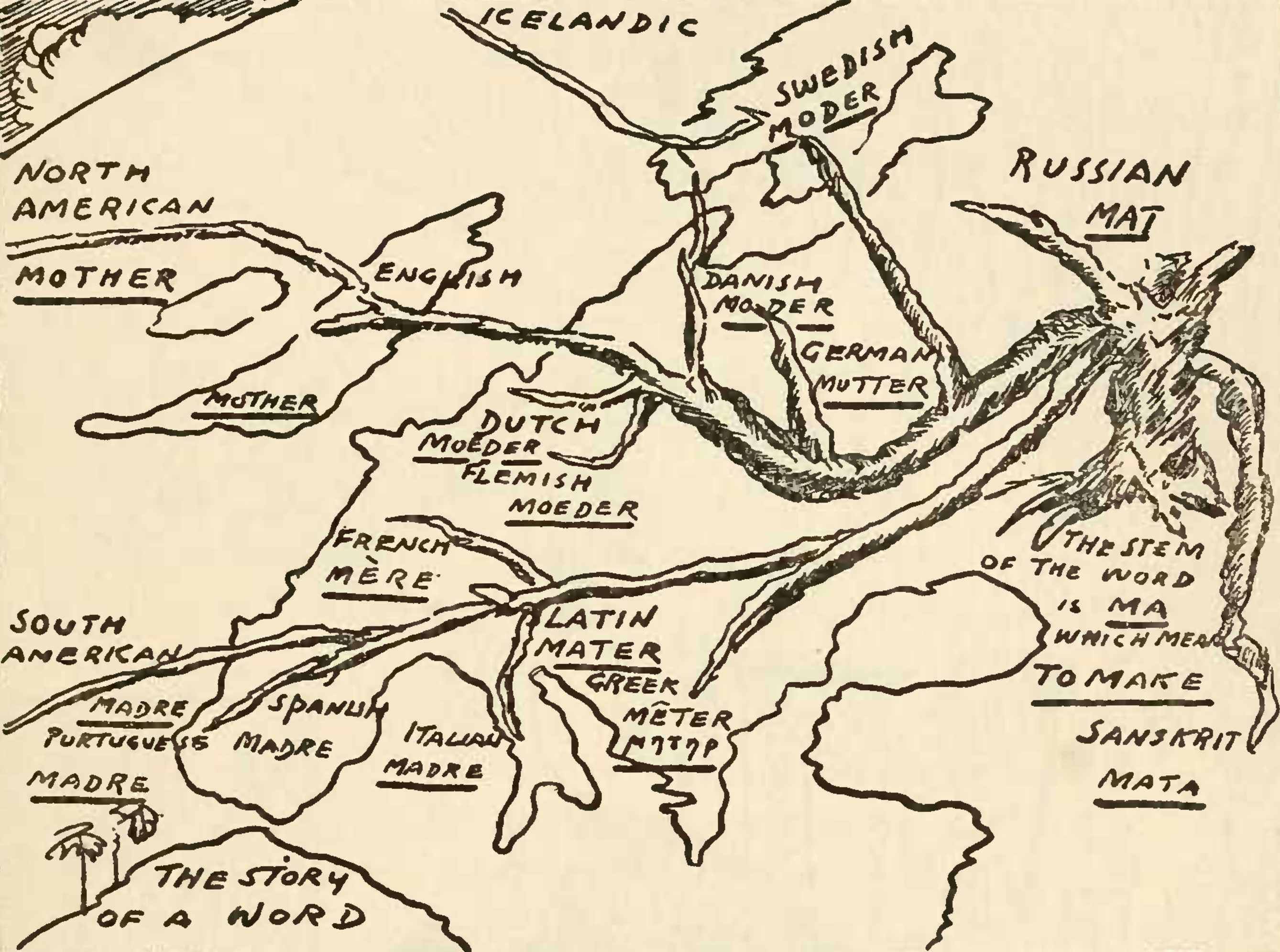 Con el DNA se puede hacer lo mismo, comparar cómo suenan las palabras (comparando letra a letra de la secuencia de genes), cómo se "conjugan" estos genes (comparando su estructura de intrones y exones), cómo se construyen las frases (comparando las secuencias reguladoras, promotores o los conjuntos de estos genes), para extrapolar la historia evolutiva de las especies que "hablan" cada DNA.
Con el DNA se puede hacer lo mismo, comparar cómo suenan las palabras (comparando letra a letra de la secuencia de genes), cómo se "conjugan" estos genes (comparando su estructura de intrones y exones), cómo se construyen las frases (comparando las secuencias reguladoras, promotores o los conjuntos de estos genes), para extrapolar la historia evolutiva de las especies que "hablan" cada DNA.
Y esto es lo que han hecho los investigadores autríaco-italianos, encabezados por Erwin Tschachler (el último firmante, el primero es Leopold Eckhart, algún día tendremos que explicar cómo funcionan los artículos científicos), comparar las secuencias de diferentes especies vivas para ver qué similitudes y diferencias presentan a nivel de los "pelos".
Los pelos están mayoritariamente formados por las proteínas alfa-queratina. Al ser proteínas, están codificadas por genes, palabras, frases comparables del DNA. Pero para poder comparar esta frase en diferentes organismos, lo primero que tenemos que saber es si tienen genes similares. Está claro que todos los mamíferos tenemos estos genes... como no podemos hacer análisis genéticos a terápsidos extintos, tendremos que tirar del hilo filogenético en busca de nuestros "primos" evolutivos más cercanos.
Los terápsidos, nuestros retataraterapsidoabuelos, se encuentran dentro de la clase de los Synapsida. Esta clase tiene una clase "hermana" que es la de los Sauropsida. Dentro de esta clase encontramos a todos los reptiles vivos actuales... y a las aves (recordad que las aves provienen evolutivamente de una parte de los dinosaurios). Los autores, por tanto, empezaron su particular búsqueda de genes en los genomas de un reptil (el anolis verde, Anolis carolinensis) y del pollo (Gallus gallus). ¿Pollo? ¿Alguien ha dicho pollo? ¿Por qué estos dos precisamente? Porque son dos de los organismos que tienen secuenciado todo su genoma (disponibles para todo el mundo en la página del PubMed, aquí). De hecho, gracias a que estos genomas (junto con otros muchos) son de dominio público la primera parte del artículo (buscar los genes de las alfa-queratinas y compararlas) las podría haber hecho cualquiera de nosotros. Pero, claro, primero se nos tendría que haber ocurrido...
Sus búsquedas les llevaron a encontrar seis genes codificantes para alfa-queratinas en el anolis, y sólo uno en el pollo. Estos genes, además, están emparentados con los genes de las alfa-queratinas de los mamíferos. Si tanto los Synapsida (mamíferos), como los Sauripsida (reptiles actuales y aves) presentan estos genes en su DNA, significa que estos genes ya se encontraban en un antepasado común. ¿En cuál? Si tiramos un poco más del hilo evolutivo la siguiente clase "hermana" la constituye la de los anfibios... Y, según los autores, éstos no tienen genes de alfa-queratina. El siguiente paso atrás, los peces, tampoco.
Todos estos datos dibujan un nuevo escenario para la aparición de los genes que forman nuestros pelos. Los genes de la alfa-queratina no son exclusivos de los mamíferos. Aparecieron en algún momento temprano de la historia evolutiva de los reptiles, y estarían presentes en los terápsidos. En el camino evolutivo hacia las aves se fueron perdiendo parte de estos genes.
A partir de aquí el artículo se complementa con un estudio de expresión de estos genes en diferentes tejidos (dónde y cuándo se expresan), mediante estudios de RT-PCR e inmunolocalización. De acuerdo, para hacer ésto sí se necesita un laboratorio, pero para la primera parte del artículo, basta un ordenador. Tengo la sensación que muchas veces se piden estos últimos resultados "de laboratorio" para evitar que cualquiera pueda publicar, porque ésta es la conclusión que nos tiene que quedar de este artículo: por un pelo no hemos publicado cualquiera de nosotros un artículo similar... sólo nos hace falta encontrar un gen interesante y realizar una búsqueda del mismo en otros organismos mediante las herramientas gratuitas del PubMed.
Mi imaginación empieza a volar y veo un futuro en el que habrá una "revista" especializada en las búsquedas y comparaciones de genes en la que todo el mundo podrá publicar... quizás más que una revista, sería un blog abierto (tipo el cedazo), con un tutorial de cómo realizar las búsquedas y comparativas. Hay tantos genes y genomas... Yes we can! Perdón me dejo llevar.
¿Alguien propone la primera búsqueda de la era democratagenómica?
Para más información sobre la historia evolutiva de los mamíferos os recomiendo este artículo de Murphy, WJ, et al. Genome Res. 2007. 17: 413-421
Images:
1. Theriognathus by DiBgd. Wikimedia commons.
2. Styracocephalus, by Karkemish. Wikimedia commons
3. Thrinaxodon fossil, by Esv. Wikimedia commons
4. The story of the word mother, by Hendrik Willem van Loon. Wikimedia commons
5. Cutreárbolgenealógico. Cosecha propia.
Publicado por
Salva
en
22:10
0
comentarios
![]()
Etiquetas: Actualidad, Evolución, Genética
28/11/08
Lab Basics (8): Corta y pega: diseña tu propio clon
 A las personas fácilmente impresionables, tendentes a simplificar la complejidad de los organismos vivos con un "esto debe ser cosa de un dios que lo diseña y lo organiza todo", les debe resultar intrigante que los científicos moleculares andemos todo el rato hablando de los ácidos nucleicos y las proteínas como si fueran bolas de plastilina, o bloques de Lego, fáciles de manipular a nuestro antojo. Pero no hay que olvidar que son moléculas y que los humanos nos limitamos a utilizar propiedades químicas y leyes físicas que nos hemos encontrado dadas: es ciencia y no magia que los enlaces atómicos puedas romperse bajo ciertas condiciones y formarse en otras. Nos limitamos a aprovecharnos de ello.
A las personas fácilmente impresionables, tendentes a simplificar la complejidad de los organismos vivos con un "esto debe ser cosa de un dios que lo diseña y lo organiza todo", les debe resultar intrigante que los científicos moleculares andemos todo el rato hablando de los ácidos nucleicos y las proteínas como si fueran bolas de plastilina, o bloques de Lego, fáciles de manipular a nuestro antojo. Pero no hay que olvidar que son moléculas y que los humanos nos limitamos a utilizar propiedades químicas y leyes físicas que nos hemos encontrado dadas: es ciencia y no magia que los enlaces atómicos puedas romperse bajo ciertas condiciones y formarse en otras. Nos limitamos a aprovecharnos de ello.
En 1975, Nathans y Smith descubrieron que las bacterias producían unas enzimas muy curiosas para protegerse del ADN extraño (viral) susceptible de atacar su material genético: las endonucleasas o enzimas de restricción. Estas proteínas actúan como auténticas “tijeras moleculares”, capaces de cortar el esqueleto fosfato de la doble cadena de ADN sin dañar las bases nitrogenadas. Premio Nobel al canto: la ingeniería genética estaba en marcha, más conocida como "esto lo quito de aquí para ponerlo allá porque me interesa más", o, para los respetuosos de las jerarquías divinas, jugar a ser Dios.
Las enzimas de restricción son proteínas que pueden ser purificadas de cultivos a nivel industrial de las bacterias que las producen, y que dan nombre a la enzima en cuestión: EcoRI (E. coli cepa RI) por poner un ejemplo arquetípico. Hay cientos de ellas comercializadas con precios variables (de 20 euros a 200 en función de la rareza y concentración). El ADN bacteriano está protegido de sus propias enzimas por presentar grupos metilo (-CH3).
Las diferentes enzimas de restricción no cortan el ADN al azar, sino porque se unen a él tras reconocer una secuencia concreta (diana de restricción). De lo contrario no cortan nada. Las dianas suelen ser secuencias cortitas y los trozos de ADN resultantes pueden tener extremos romos o con fragmentos de secuencias protuberantes. Un extremo protuberante de una cadena de ADN "A" puede unirse a un extremo protuberante con secuencia complementaria de la cadena de ADN "B", que es la gracia del tema. Las enzimas requieren para actuar unas condiciones especiales, para lo cual el proceso de digestión que tiene lugar en el tubo debe darse poniendo en contacto el ADN que se desea cortar con las enzimas de interés, un tampón con iones apropiados y la temperatura idónea apara la enzima (habitualmente, 37ºC), y dejar que las tijeras corten durante 1 hora o más horas.
Las enzimas requieren para actuar unas condiciones especiales, para lo cual el proceso de digestión que tiene lugar en el tubo debe darse poniendo en contacto el ADN que se desea cortar con las enzimas de interés, un tampón con iones apropiados y la temperatura idónea apara la enzima (habitualmente, 37ºC), y dejar que las tijeras corten durante 1 hora o más horas.
Un batallón de otras proteínas colaboran en los procesos de ingeniería genética: ligasas (el pegamento necesario para que los fragmentos de ADN cortados puedan volverse a unir), polimerasas especiales que cortan extremos protuberantes y los vuelven romos, otras proteínas que impidan que un fragmento digerido vuelva a unirse solo...Un maletín de manualidades puesto al servicio de tu propio diseño de material genético.
Podéis entreteneros comprobando que cualquier secuencia es susceptible de contener dianas de restricción inventándoos una secuencia en un buscador de dianas aquí.
De lo que hemos aprendido en las entradas anteriores de la serie podemos deducir cuales son los procedimientos habituales: se purifican los ADN de interés, se amplifica la región concreta que deseemos cortar y pegar; los fragmentos de ADN digeridos pueden diferenciarse por electroforesis; podemos purificar ese ADN y disponer de su secuencia para buscar las dianas de restricción que contenga y elegir las enzimas de restricción adecuadas. Cortamos los fragmentos deseados y los podemos unir entre sí, uniendo moléculas de ADN de distinta procedencia. Pegamoslos fragmentos y ese ADN se puede introducir en una célula para que se replique normalmente (bacteriana o eucariota dependiendo de las modificaciones que después queramos que tenga la proteína).
Lo más habitual es que las víctimas de todos esos procesos sean las propias bacterias. A una bacteria a la cual se le ha introducido un fragmento de material genético procedente de otro organismo se le llama "clon" (a distinguir de lo que la mayoría de la gente entiende por "clonación"). A nivel comercial disponemos de cromosomas bacterianos circulares modificados (vectores) que contienen una región rica en dianas de restricción para que sea fácil cortar y pegar nuestro gen de interés, con regiones que permitan que ese gen se exprese en bacterias o células eucariotas, y otro gen que produce una proteína con propiedades antibióticas de manera que podamos matar las bacterias que no han introducido ese vector clonado y nos quedemos sólo con las amables bacterias que contienen nuestro vector y resisten al antibiótico, aparte de contener nuestro gen y/o producir nuestra proteína de interés.
También podemos cortar y pegar nuestro gen al gen que codifique una proteína fluorescente de manera que cuando se fabrique nuestra proteína, lleve colgando un farol luminoso que nos indique dónde está (esto lo veremos en una entrada posterior).
La lista de aplicaciones industriales, farmacéuticas y científicas es enorme: produccion de anticuerpos, proteínas de interés humano (producción de insulina para diabéticos, por ejemplo), generación de vacunas (como la de la hepatitis), modificación genética de plantas y animales (¡¡es la base de los transgénicos!!), terapia génica, y aplicaciones en medicina forense.
No obstante, la producción industrial por bacterias modificadas genéticamente de moléculas para consumo humano tiene muy mala prensa a raíz del trágico caso Showa Denko, en el que a finales de los 80 murieron 38 personas y más de 1500 quedaron seriamente afectadas por consumo de un suplemento dietético (L-triptófano) producido por bacterias modificadas al que no se le realizaron los debidos controles sanitarios; los grupos contrarios a la comercialización de alimentos transgénicos lo tienen como ejemplo preferido y más letal de los peligros de la manipulación genética. Vamos, es que no hay muchos más que hayan sido letales...Y no, la enfermedad de las vacas locas no es un ejemplo de efectos nocivos derivados de la manipulación genética, porque es una enfermedad infecciosa, producida por priones.
Publicado por
Elena Garrido
en
9:40
0
comentarios
![]()
Etiquetas: Series divulgativas
En el (ADS)L: Entradas interes(ADN)tes, 28-11-08
- Educación y ciencia, en Evolucionarios. Absolutamente genial. Imprescindible. Un buen análisis de la situación de las carreras científicas en España. Los porqués. Los quéhacer... Vale mucho la pena.
También os recomendamos...
- ¿La primera familia nuclear?, en La lógica del Titiritero. Buena entrada sobre la "mediática" noticia referente al descubrimiento de una tumba neolítica con cuatro individuos del mismo núcleo familiar y su tratamiento en los medios.
- Más fósiles transicionales de tortugas: Odontochelys, en Paleofreak. De la adquisición o no del caparazón... ¿Fósiles transicionales? Ves preparando el cheque, creacionista turco.
- El atractivo de los Harrijasotzaileak (o ¿por qué levantan piedras algunas aves?), en El cerebro de Darwin. Interesante entrada sobre aves y piedras, sin pasar por ninguna molleja... Empezaré a llevar piedras de 20 kilos por la calle, a ver qué pasa.
- ¿En qué consiste la vacuna española contra el VIH?, en Museo de la Ciencia... y es que se están convirtiendo en fijos de esta sección. Ya que estoy, os recomiendo también Caballos de Troya moleculares.
- Y en el apartado de humor, esta entrada de Ciencia de la Vida: Viñetas sobre la educación. Mejor tomárselo a broma.
Publicado por
Salva
en
7:14
2
comentarios
![]()
Etiquetas: Actualidad, Ciencia en los medios
25/11/08
La ciencia de Second Life
Hace poco, un muy buen amigo -saluda, David- me envió esta noticia de Javier Martín de El País, en la que se habla sobre la utilización de los videojuegos -y la informática en general- en la educación. El tema ya sería interesante de por sí, pero lo es más aún si tenemos en cuenta que algunos de nosotros nos dedicamos a realizar libros de texto completamente digitales para la secundaria...
Deformaciones laborales a parte, en el artículo se hablaba de la utilización de algunos "juegos" en determinadas investigaciones científicas. Y ponían de ejemplo Second Life. ¿Ah si? ¿Hay artículos de investigación en los que se utilice este juego de realidad virtual? Mmmmmm. Sentía que debía acudir a mi querido buscador de artículos biomédicos Pubmed. Y he aquí lo que hallé...
Básicamente, los artículos en los que aparecía "Second life" podrían agruparse en:
Entradas relacionadas: Viciats però normals, quin descans d'Abulafia (en catalán)
Publicado por
Salva
en
13:41
0
comentarios
![]()
Etiquetas: Actualidad, Antropología, Ciencia en los medios
Prescribe Ciencia 2008: Educación y divulgación por internet
Este jueves 27 de noviembre a las 19:00, en la sede del Instituto Cervantes de Madrid (c/ Barquillo, 4), tendrá lugar el encuentro que tratará sobre La Ciencia en Internet (podéis consultar el calendario completo de actos aquí).
Enterados a través de la ACCC
Publicado por
Salva
en
6:00
0
comentarios
![]()
Etiquetas: Actividades, Actualidad
23/11/08
Gusiluz, el molusco fotosintético y la transferencia horizontal de genes
 Uno de los temas más utilizados en los argumentos de novelas, cómics, videojuegos o películas de aventuras es el del equipo de especialistas. Desde El Señor de los Anillos, las crónicas de la Dragonlance, la serie de Comandos, hasta los cómics de El Jabato o El Capitán Trueno (¿o era el mismo?), se han constituido equipos, grupos, hermandades, o como se les haya querido denominar, de personajes que, juntos, generan un "ente" muy superior a la suma de sus habilidades. Enanos, elfos, humanos y hobbits; arqueros, guerreros, magos y sanadores; francotirador, espía, cepador...
Uno de los temas más utilizados en los argumentos de novelas, cómics, videojuegos o películas de aventuras es el del equipo de especialistas. Desde El Señor de los Anillos, las crónicas de la Dragonlance, la serie de Comandos, hasta los cómics de El Jabato o El Capitán Trueno (¿o era el mismo?), se han constituido equipos, grupos, hermandades, o como se les haya querido denominar, de personajes que, juntos, generan un "ente" muy superior a la suma de sus habilidades. Enanos, elfos, humanos y hobbits; arqueros, guerreros, magos y sanadores; francotirador, espía, cepador...
Siguiendo este "hilo argumental" en esta entrada viajaremos desde la máxima de "la unión hace la fuerza" hasta un molusco fotosintético, nuestro particular gusiluz.
En biología la máxima "1+1 suman más que dos" se ha seguido en muchas ocasiones. Los líquenes son asociaciones de algas con hongos que producen algo nuevo. Las sociedades de insectos, con sus especializaciones constituyen verdaderos supra-organismos. Los mismos organismos pluricelulares somos sociedades de millones de células especializadas.
Las propias células eucariotas son asociaciones duraderas de diferentes bacterias (es la famosa teoría endosimbiótica). ¿Cómo se produjo esta asociación? ¿Cómo bacterias tan diferentes (la proto-célula nucleada y los precursores de las mitocondrias y cloroplastos) acabaron juntas? Una de las teorías que se barajan es la de la "mala digestión" en la que nos encontraríamos una bacteria grande que se "tragaría" otras bacterias sin digerirlas del todo. Esta asociación se adaptaría mejor al medio que le habría tocado vivir y fue seleccionada positivamente dando lugar a las células eucariotas actuales
En el último número del PNAS (Proceedings of the National Academy of Science) se publica un artículo (Firmado en primer lugar por Mary Rumpho, y editado por Lynn Margulis) centrado en el estudio del molusco fotosintético Elysia chlorotica, una babosa marina de color esmeralda.
Los animales, hongos, algas eucariotas y vegetales son todos organismos eucariotas porque están formados por las células eucariotas. Todas las células de estos supergrupos tienen núcleo y mitocondrias; las células de las algas eucariotas y los vegetales son las únicas que tienen cloroplastos. Los cloroplastos son los orgánulos que realizan la fotosíntesis transformando el dióxido de carbono y el agua en glucosa (el combustible de los seres vivos) gracias a la energía que le aporta la luz solar. Los cloroplastos, llenos, entre otras, de clrofila, son los que le dan el color verde a las plantas... y a la babosa marina Elysia chlorotica (que no es lo mismo que decir la babosa del Elíseo, que nadie piense mal. Quelqu'un m'a dit).
Un momento, un momento, no nos desviemos... ¿no decíamos que eran exclusivos de algas aucariotas y vegetales? Y así es. Esta especie de babosa ha coevolucionado con una especie de alga pudiendo "adoptar" sus cloroplastos para su uso y disfrute.
Cuando una nueva babosa nace, lo hace sin cloroplastos... a lo largo de su vida los va adquiriendo. ¿De dónde? Entre los platos preferidos del gusiluz se encuentra la alga eucariota Vaucheria litorea. Cuando un gusiluz come estas algas no digiere todos sus cloroplastos. Algunos se incorporan a las células de su sistema digestivo. Y allí siguen su tarea fotosintética, produciendo glucosa para el gusiluz.
Pero, ¿es esto así de sencillo? En un principio lo parece. Esa alga tiene algo (perdón) que le produce glucosa. Me la como, se lo quito y me lo quedo. ¿Por qué no hacemos nosotros lo mismo? Evidentemente, la cosa no es tan banal.
Durante los cientos de millones de años que llevan coevolucionando los orgánulos de las células eucariotas se han establecido relaciones que aseguran su funcionamiento como un todo, pero que hacen que los componentes de estas células, pese a que antes eran bacterias de vida libre, ahora no puedan sobrevivir por separado. Una mitocondria ya no puede sobrevivir sin la célula eucariota ya que algunos de los genes que codifican para proteínas de la mitocondria se encuentran ahora en el núcleo. Sin el núcleo, las mitocondrias no están completas. Y lo mismo pasa con los cloroplastos. Los cloroplastos de las algas, incluídos los de la Vaucheria litorea, son incapaces de sobrevivir y funcionar correctamente sin las proteínas codificadas en genes que se encuentran en el núcleo de las algas (como la proteína psbO) .
Y aquí viene la novedad del trabajo que hoy comentamos. Los cloroplastos de la Vaucheria sobreviven y funcionan dentro de las células de la babosa Elysia pese a que no tienen "cerca" el núcleo de su alga. Si necesitan las proteínas codificadas por el ADN de la alga... ¿cómo funcionan en la babosa? Porque el núcleo de la babosa también codifica para estas proteínas (al menos, para la psbO). Es decir, en el DNA de todas las babosas de la especie E. chlorotica encontramos genes originarios de algas (como se demuestra al comparar la secuencia de los genes de las dos especies).
Es evidente que las babosas y las algas han seguido caminos evolutivos muy y muy distintos desde que se separaron de su ancestro común y que los genes que las algas adquirieron durante sus miles de millones de años de convivencia con los cloroplastos son totalmente ajenos a los animales, grupo al que pertenecen las babosas. ¿Cómo han podido adquirir este gen las babosas? Mediante lo que se conoce como Transferencia Horizontal de Genes.
La transferencia vertical es la que se da de padres a hijos y sigue la línea de la historia evolutiva de cada una de las especies. La horizontal se da entre diferentes especies. Esta transferencia se produce sobre todo entre organismos procariotas (las resistencias de determinadas bacterias a antibióticos siguen a veces esta vía); es rara entre procariotas y eucariotas (como el que se da entre el procariota edosimbionte Wolbachia y las células de sus huéspedes insectos y nemátodos); y es prácticamente inexistente entre organismos eucariotas (hasta el momento se han descrito como anecdóticas transferencias entre plantas parásitas y las plantas parasitadas, transferencia de transposones, o transferencias entre las plantas y nemátodos parasitarios). La probabilidad de que se de esta transferencia entre células nucleadas es tan baja que seguramente es necesaria una relación de millones de años entre las especies para que esta transferencia se estabilice, como en el caso de nuestro gusiluz y su plato favorito.
Quedan, como siempre, muchos interrogantes que resolver. Entre ellos, ¿cómo se produjo la integración del gen en el genoma? ¿Recombinación? ¿Virus? ¿Retrovirus? ¿Cuánto hace de esta integración? ¿Qué otros genes se han integrado? ¿Podría ser esta una relación que fuese a más, es decir, que en algún punto las babosas ya naciesen con los cloroplastos? ¿sería entonces una nueva especie? ¿una nueva familia? ¿orden?
Si pensamos ahora en la cantidad de especies que existen sobre la Tierra, en las relaciones que se establecen entre ellas y en la mejora de los mecanismos para secuenciar genomas (nucleares, mitocondriales o de cloroplastos)... ¡cuántas relaciones nuevas nos quedan por descubrir!
Publicado por
Salva
en
18:00
2
comentarios
![]()
Etiquetas: Actualidad, Genética
ADSADN va a la radio
Este martes ADSADN va a la radio. Concretamente al programa Calaix d'Arquimedes (del que llevamos tiempo recomendandoos su blogen catalán) de Ràdio Mataró, de 17:00 a 18:00...
Veremos cómo nos va...
Publicado por
Así de Natural
en
14:52
2
comentarios
![]()
Etiquetas: General
21/11/08
En el (ADS)L: Entradas interes(ADN)tes, 21-11-08
Esta semana hemos leído y os recomendamos:
Otras entradas recomendadas...
Publicado por
Salva
en
9:12
0
comentarios
![]()
Etiquetas: Ciencia en los medios
19/11/08
Amusia: Desórdenes musicales
Me estoy leyendo el DEA de un compañero de trabajo, Oriol Solé, sobre la relación del lenguaje, la música y el cerebro. Un texto muy interesante, la verdad, del que tengo ganas de hablaros con más calma. De momento, os dejo una pequeña perla, de la que no tenía ninguna noticia: las amusias.
Los pacientes que presentan amusia (un 4% de la población>) son incapaces de reconocer o reproducir o tonos o ritmos; otros pueden tener dificultades con el lenguaje musical escrito. No me ha dejado de parecer curiosa la existencia de estos síndromes, y no sólo por su "peculiaridad", sino también por lo que representan, ya que son síndromes causados por afectaciones en distintas áreas cerebrales: es decir, hay áreas del cerebro especializadas en cada una de estas características de la música. Como el lenguaje.
Ya conocéis mi pequeña obsesión por consultar en el PubMed todo lo que puedo (bien sean GFPs, ojeras, o básquet). En esta ocasión tampoco me he podido controlar y, como de costumbre, la búsqueda me ha ofrecido resultados interesantes:
Publicado por
Salva
en
21:32
0
comentarios
![]()
Etiquetas: Curiosidades, General, Neurobiología
18/11/08
Premios 20blogs
Bueno, ya se ha finalizado la edición de este año de los premios 20blogs.
Antes de nada, queremos felicitar al ganador de la categoría de Ciencia y medio ambiente: La lógica del titiritero.
Nuestro blog ha quedado en la posición 87... ¡con 1 voto! ¡Gracias Tito! No sabes lo que significa tener un voto en una edición como ésta... bueno, ¡sí lo sabes! La caricatura existencialista tiene 4.. ¡felicidades!
Y de cara al año que viene, a mejorar los resultados. ¡Claro que sí!
Publicado por
Así de Natural
en
8:00
0
comentarios
![]()
Etiquetas: Actualidad, General
16/11/08
¿Desde cuándo los humanos hablamos (de ciencia)?
Hoy quiero hablaros de un libro que tengo desde hace tiempo pero que otras lecturas fueron atrasando la suya. Me refiero a Hablemos de Ciencia de José Manuel Nieves. Aprovecharé, además, para comentaros un artículo comentado al final del libro sobre cómo estudiar si los homínidos ya extintos hablaban o no. PEro empecemos por el libro...
El libro: Hablemos de Ciencia Este libro es el primero de la colección de divulgación científica Conversaciones de Ciencia de EDAF, cuyo catálogo podéis consultar aquí.
Este libro es el primero de la colección de divulgación científica Conversaciones de Ciencia de EDAF, cuyo catálogo podéis consultar aquí.
En el libro de José Manuel Nieves nos encontramos con un repaso por diferentes áreas de las Ciencias (Universo, Estrellas, la Tierra, la Materia, la Vida y el Hombre). El libro está escrito en un estilo altamente accesible, profusamente ilustrado con fotografías y gráficos, diferente a lo que solemos entender como libros de divulgación (al menos los no anglosajones). Además, se añaden explicaciones de actualidad en cada uno de los campos mencionados.
La verdad es que con una presentación así, me han entrado ganas de conseguir alguno de los otros libros de la colección y ver si logran mantener el nivel que éste ha marcado. Porque si es así, valdrán mucho la pena.
El artículo: Homo heidelbergensis, Allo? Señorita?
En el apartado de el Hombre, José Manuel Nieves, nos sitúa en el punt en qué se encuentra uno de los debates más candentes de la evolución humana... ¿en qué punto empezaron los humanos a hablar? ¿Es una característica exclusiva de los H. sapiens? Si no es así, ¿qué otras especies de Homo la presentaban? La cuestión no es baladí, ya que los indicadores de un lenguaje articulado no fosilizan con facilidad. Se ha intentado abordar el estudio de esta cuestión desde estimaciones del tamaño de las zonas del cerebro responsables en la interpretación del lenguaje, pasando por la estimación de la posición del aparato fonador en la laringe... El artículo del que quiero hablaros proponía un nuevo enfoque a este problema y apareció en el PNAS del 20 de mayo de 2004, o sea, que se puede empezar a considerar "un clásico", y fue desarrollado y firmado por el equipo de Atapuerca (I. Martínez, M. Rosa, J.-L. Arsuaga, P. Jarabo, R. Quam, C. Lorenzo, A. Gracia, J.-M. Carretero, J.-M. Bermúdez de Castro, and E. Carbonell).
La cuestión no es baladí, ya que los indicadores de un lenguaje articulado no fosilizan con facilidad. Se ha intentado abordar el estudio de esta cuestión desde estimaciones del tamaño de las zonas del cerebro responsables en la interpretación del lenguaje, pasando por la estimación de la posición del aparato fonador en la laringe... El artículo del que quiero hablaros proponía un nuevo enfoque a este problema y apareció en el PNAS del 20 de mayo de 2004, o sea, que se puede empezar a considerar "un clásico", y fue desarrollado y firmado por el equipo de Atapuerca (I. Martínez, M. Rosa, J.-L. Arsuaga, P. Jarabo, R. Quam, C. Lorenzo, A. Gracia, J.-M. Carretero, J.-M. Bermúdez de Castro, and E. Carbonell).
En este trabajo se decidió no mirar al emisor sino al receptor. ¿De qué sirve que alguien hable si nadie le escucha? Los humanos tenemos los huesos de nuestro oído interno "sintonizados" para captar frecuencias entre 1 y 4 kHz, las frecuencias en las que se mueve el habla. Los chimpancés tienen sus oídos "sintonizados" en frecuencias cercanas o bien a 1 kHz o bien a 8 kHz. Contando con los resultados de estudios de medidas físicas de los huesos de estas especies se pudo inferir a qué frecuencias estaban adaptados los oídos de los cráneos de Homo heidelbergensis encontrados en la Sima de los Huesos (Atapuerca). Y éstos presentaban una buena "afinación" a 2-4 kHz. ¿Se hablaban entre ellos? Lo que es seguro es que podían oirse si lo hacían.
Los Homo heidelbergensis se sitúan en el camino evolutivo que lleva a los neandertales. De este hecho, por tanto, podríamos deducir un par de cosillas interesantes:
Particularmente me estremece la idea de que durante unos milenios convivieron diferentes especies de humanos hablándose entre ellos, comunicándose, intercambiando materiales, productos, ideas, temores... ¿es una imagen demasiado bucólica? ¿Creéis que los sapiens atacarían antes de preguntar? ¿O preguntarían antes de... qué? ¿A quién se lo preguntamos?
Tantos oídos para que no haya habido una infinidad de palabras
Publicado por
Salva
en
11:00
0
comentarios
![]()
Etiquetas: Antropología, Evolución, Lecturas recomendadas





